- Startseite
- Forschung
- Translationale Forschung
- AG Computational Surgical Oncology
AG Computational Surgical Oncology
English version
Die Arbeitsgruppe Computational Surgical Oncology führt translationale Forschung auf dem Gebiet der Künstlichen Intelligenz und der Datenwissenschaft (data science) durch. Kernkompetenz ist die sogenannte Computer Vision, bei der Bilder als Eingabe für die neuronalen Netze verwendet werden. Dabei werden Techniken des “deep learnings” angewendet. Zum Einsatz kommen vor allem Convolutional Neural Networks (CNNs), die im Bereich der onkologischen Chirurgie, Onkologie und Chirurgie eingesetzt werden. Mit diesem Ansatz wird die personalisierte Medizin vorangetrieben. Besonders die personalisierte Krebstherapie steht hier im Fokus.
Unsere Vision
Personalisierung von Therapieentscheidungen durch Transformation aller digitalen Patientendaten in Biomarker mit der Hilfe von künstlicher Intelligenz.
Oft gibt es mehrere, gleichwertige Therapien für einen Patienten. Die personalisierte Therapie basiert auf der Fähigkeit vorherzusagen, auf welche dieser Therapien ein Patient am besten ansprechen wird. Für diese Vorhersage werden vor allem in der Krebsmedizin meist Biomarker herangezogen. Zum aktuellen Zeitpunkt stehen für individuelle Therapieentscheidungen wenige Biomarker zur Verfügung, auf deren Basis meist eine optionale, gezielte Therapie initiiert wird. Bisher werden für die Individualisierung von Therapieentscheidungen wenige Biomarker gemessen, auf deren Basis meist eine optionale, gezielte Therapie initiiert wird.
Ein Beispiel für solch einen Biomarker mit dem gezielt das Ösophaguskarzinom therapiert werden kann, ist der humane epidermale Wachstumsfaktor-Rezeptor 2 (HER2). HER2, ein Mitglied der EGFR-Familie, ist auf der Zellmembran lokalisiert und beschleunigt das Tumorwachstum. Als Biomarker wird der HER2-Status für eine potenzielle Gabe einer Anitkörpertherapie mittels Trastuzumab bestimmt. Bei einer vorhanden HER-2 Expression, das heißt einem positiven HER–2 Status, kann Trastuzumab als Therapie beim palliativen Ösophaguskarzinom empfohlen werden.
Je mehr Biomarker verwendet werden, desto genauere Therapieentscheidungen können individuell getroffen werden. Die Idee der Arbeitsgruppe Computational Surgical Oncology ist es, alle verfügbaren Patientendaten zu digitalisieren und als Biomarker für individuelle Therapieentscheidungen heranzuziehen. Mit Hilfe von neuronalen Netzwerken können die dabei entstehenden extrem komplexen und umfangreichen Daten beherrscht werden und in individuelle Therapieentscheidungen transformiert werden.
Besonders interessant sind pathologische Bilddaten, wie morphologische H&E-Färbungen, die mehr Informationen enthalten, als für das bloße Auge sichtbar sind. So ist es gelungen, molekulare Mutationen anhand von H&E-Daten vorherzusagen. Die Arbeitsgruppe Computational Surgical Oncology benutzt digitale H&E-Färbungen (ca. 10 Gb pro Bild), um das Ansprechen auf Krebstherapien individuell vorherzusagen. In Zukunft kann so individuell entschieden werden, ob ein Patient sofort operiert oder besser eine neoadjuvante Therapie erhalten soll.
Die Art und Anzahl der Biomarker wird für die Personalisierung der Krebstherapie schrittweise erhöht. Mit den H&E-Färbungen werden klinische Basisdaten kombiniert, um die Vorhersage des Ansprechens auf eine Therapie zu optimieren. Später wird auch die radiologische Bildgebung wie zum Beispiel die Computertomographie (CT) als Biomarker integriert werden. Neben der Wahl der optimalen individuellen Therapie kann mithilfe von Methoden der künstlichen Intelligenz individuell die Prognose bei einer Krebserkrankung bestimmt werden.
Mithilfe von digitalen Biomarkern können wichtige chirurgische Entscheidungen bei Operationen unterstützt werden. Neben der Krebstherapie liegt der Fokus der Arbeitsgruppe Computational Surgical Oncology in der AI-gestützten Optimierung der Operationsstrategie. Biomarker können beispielsweise bei der Planung der Operation helfen, das für einen Patienten individuell geeignetste Operationsverfahren auszuwählen. Methoden der künstlichen Intelligenz helfen, das Operationsrisiko individuell abzuschätzen.
Laufende Projekte
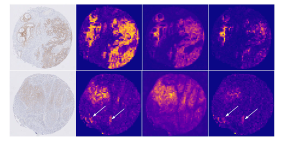
Vorhersage des HER2-Status beim Ösophaguskarzinom aus Tissue Mikroarrays mit Hilfe von neuronalen Netzen
Unser Ziel ist es, eine Deep-Learning-Methode zu entwickeln, mit der mikroskopische Bilder von Ösophaguskarzinomen auf das Vorhandensein einer HER2-Überexpression untersucht werden können. Unsere Lösung ermöglicht den direkten automatischen Nachweis von HER2 in immunhistochemisch gefärbten Gewebeschnitten, ohne dass eine manuelle Bewertung und zusätzliche kostspielige In-situ-Hybridisierungstests (ISH) erforderlich sind.
Wir haben neuronale Netze (CNNs) auf einem umfangreichen Datensatz von 1.602 Patientenproben trainiert und an einem unabhängigen Satz von 307 Patientenproben getestet. Dabei haben wir einen Aufmerksamkeitsmechanismus in die CNN-Architektur eingebaut, um die Geweberegionen zu identifizieren, die das Netzwerk als wichtig die Vorhersage erkennt.
Unsere Methode weist eine Genauigkeit von 0,94, eine Präzision von 0,97 und eine Wiedererkennung von 0,95 auf. Wichtig ist, dass unser Ansatz genaue Vorhersagen in Fällen bietet, die von Pathologen nicht geklärt werden können und zusätzliche ISH-Tests erfordern.



